1.1.3. LES BIOPUCES
1.1.3.1. Les technologies utilisées
L'hybridation : théorie
Le
concept de biopuce ou puce à ADN date du début des années
1990. Il a été à cette époque décrit par
quatre équipes différentes, en Grande-Bretagne, en Russie et aux
États-Unis. Il repose sur une technologie pluridisciplinaire
intégrant la micro-électronique, la chimie des acides
nucléiques, l'analyse d'images et la bio-informatique. Le principe de
fonctionnement de ces puces repose sur un fondement de la biologie
moléculaire : le phénomène de l'hybridation,
c'est-à-dire l'
appariement
par complémentarité
des bases de deux séquences d'ADN
. Par analogie, on peut comparer ce
mécanisme à celui de la fermeture éclair... En effet, les
brins extraits de la double hélice d'ADN ont la capacité de
reformer spontanément cette double hélice dès qu'ils se
retrouvent face au brin complémentaire ; les quatre
molécules élémentaires de l'ADN ont la
particularité de s'unir deux à deux :
l'
adénine
avec la thymine
,
la cytosine
avec la
guanine
.
Le phénomène de l'hybridation permet d'identifier une
séquence de nucléotides, c'est-à-dire l'enchaînement
des bases d'un fragment d'ADN en mettant ce dernier en présence d'autres
brins d'ADN, dont la séquence est connue.
Par exemple, face à des brins d'ADN synthétique
représentatifs d'une maladie, les brins extraits de l'ADN du patient
vont hybrider si le malade est porteur de l'affection recherchée.
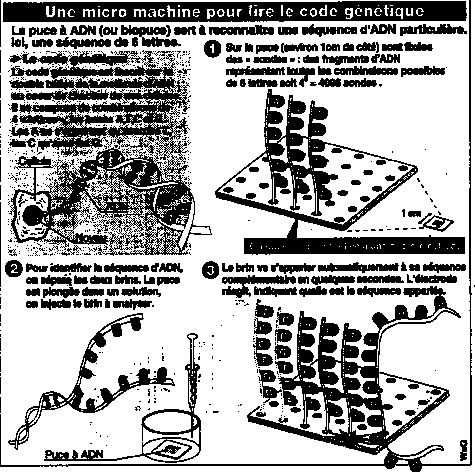
Le problème est " d'offrir " aux brins d'ADN du patient la possibilité d'hybrider avec de nombreux brins d'ADN synthétiques représentant les différents gènes correspondant à une pathologie, sous leurs différentes formes.
Le support
Les
brins synthétiques utilisés, ceux dont on connaît la
séquence, s'appellent des sondes. Les puces à ADN
présentent, par rapport aux méthodes classiques d'hybridation un
avantage majeur : grâce aux techniques de miniaturisation elles
permettent l'hybridation simultanée d'un nombre de sondes
considérables sur une surface totale utile inférieure à
1 cm
2
. Les supports sur lesquels sont fixées les sondes
se présentent sous la forme de surfaces planes ou poreuses
(percées de puits) composés de matériaux tels que :
-
le verre
, matériau peu onéreux, inerte et
mécaniquement stable. Il est notamment utilisé par l'entreprise
américaine Affymetrix et le consortium Genosensor). La
société américaine Protogene (Palo Alto, Californie) avec
laquelle les chercheurs de l'équipe du Professeur Francis GALIBERT, du
CNRS, ont passé des accords de partenariat utilise une ingénieuse
technique : la petite plaque de verre, substrat de la puce, est recouverte
d'une grille de Teflon (qui détermine des zones hydrophiles et
hydrophobes).
-
les polymères
sont à la base des techniques
développées par l'équipe d'Andrei MIRZABEKOV (Argonne,
Illinois) ainsi que par les chercheurs de la société CIS
Bio-international qui utilisent plus précisément le
polypyrrole ;
-
le silicium
, couramment utilisé pour la fabrication des
puces électroniques à cause de ses propriétés
semi-conductrices (matériau choisi par plusieurs équipes et
notamment par le laboratoire IFOS de l'École centrale de Lyon -
UMR 5621 du CNRS).
- les métaux, notamment
l'or et le platine
(utilisés
par la société américaine Nanogen et le Consortium
Genosensor).
La fixation des sondes
Quel que
soit le support choisi, il est traité pour former un réseau dense
et régulier de microsurfaces et sur chacune de celles-ci est
greffée une sonde d'ADN synthétique. Il existe deux
méthodes : le transfert de brins d'ADN ou sa synthèse
in
situ
.
- La
synthèse préalable
à la fixation des sondes
permet de fixer des sondes relativement longues, atteignant 40 à
60 bases. Le
transfert
de ces sondes sur la puce peut se faire au
moyen de micropipettes, de micropointes ou par des dispositifs de type jet
d'encre.
Un autre système, choisi par la société CIS Bio
International en collaboration avec les chercheurs du CEA à Grenoble
(LETI)
15(
*
)
est l'adressage
électrochimique : la puce est composée d'un support en
silicium recouvert, à chaque plot d'une mini-électrode en or.
Chaque sonde rejoint un plot particulier lorsque les mini-électrodes
sont mises sous tension sélective.
Les méthodes d'adressage des sondes sont fondamentales : leur
performance, leur capacité à être automatisées et
leur coût de fonctionnement pèseront lourd dans le
développement de la technologie des puces à ADN.
-
La synthèse
in situ
:
dans ce cas, la
construction des sondes se fait par dépôt de couches successives
des quatre bases de l'ADN sur un support en verre. C'est un masque, dont la
configuration varie à chaque dépôt d'une couche, qui permet
aux bases de s'empiler correctement. Avec ce procédé,
utilisé par Affymetrix, les sondes comportent au maximum 30 bases.
Là encore, la société Protogene innove : elle a mis
au point un système qui permet de déposer sur la grille de
téflon de la biopuce, les quatre bases désirées,
grâce à des injecteurs piézo-électriques, selon une
technologie qui s'apparente à une synthèse d'ADN classique. C'est
l'équivalent d'une imprimante à jet d'encre à quatre
couleurs (chaque couleur étant en l'occurrence une base : A, G, T,
C). Ce procédé est extrêmement flexible : il permet de
fabriquer indifféremment telle ou telle sorte de biopuce, de même
qu'une imprimante imprime indifféremment telle ou telle page, quels que
soient les mots du texte.
L'hybridation : réalisation pratique
Le
rôle de chaque sonde est de reconnaître, dans un mélange
appliqué à la surface de la biopuce, une séquence d'ADN.
Généralement cette séquence est amplifiée par
PCR
16(
*
)
puis marquée par
fluorescence, préparée en solution et mise en contact avec la
biopuce.
La phase d'hybridation est réalisée dans une sorte d'incubateur
(station fluidique) et suivie d'un lavage destiné à
débarrasser la puce des cibles nucléiques non hybridées.
La détection des hybridations
Elle
permet de déterminer à quelle sonde s'est appariée la
séquence d'ADN analysée.
Le procédé Affymetrix repose sur l'utilisation d'un scanner qui
permet de repérer les sondes d'ADN devenues fluorescentes,
c'est-à-dire ayant donné lieu à une hybridation. D'autres
procédés sont à l'étude chez Clinical Micro Sensors
(Californie) ou à l'École centrale de Lyon et se fondent sur les
modifications des charges électriques du support en silicium où
sont fixées les sondes (système GenFet). La mesure de ces
modifications permet de repérer les associations spécifiques
entre les sondes et les oligonucléotides cibles.
1.1.3.2. Les possibilités d'utilisation
|
LES APPLICATIONS 17( * ) |
||||
|
Diagnostic |
Pharmacie |
Recherche |
Agro-alimentaire |
Environnement |
|
Immuno-essais hormone & cancer |
Criblage médicamenteux " screening " |
Séquençage, analyse de séquences |
Classification |
Microbiologie |
|
Microbiologie ADN antigènes/anticorps |
Mise au point médicaments |
Tri moléculaire |
Contamination |
Polluants |
|
Facteurs aggravants cardio-vasculaire vieillissement |
Pharmacogénomique |
Vision intégrée des voies métaboliques |
|
Plantes |
|
Toxicologie |
|
|
||
|
Aide au suivi des essais cliniques, adaptation de la thérapie |
|
|
|
|
Le séquençage par hybridation (SPH)
Alors
que la méthode enzymatique dite méthode de Sanger peut être
considérée comme une épellation de la séquence,
c'est-à-dire une lecture base par base, le SPH procède par
lecture de petits blocs. L'analyse porte sur des sous-séquences
chevauchantes qui sont lues et réassemblées au moyen d'un
programme informatique qui reconstitue la séquence étudiée.
Le séquençage par biopuces constitue une alternative
intéressante et plus précise à la méthode classique
du séquençage en terme d'automatisation et de réduction
des coûts et des durées d'exécution. Cependant tous les
problèmes techniques ne sont pas résolus.
L'identification de cibles pour la recherche thérapeutique :
La contribution des biopuces au séquençage s'accompagne d'une aide à la compréhension plus fine du génome et de sa régulation. Ainsi, grâce aux puces à ADN, ont été mis en évidence de nouveaux gènes s'exprimant spécifiquement dans le tissu cérébral de l'enfant, ou apparaissant associés à des pathologies inflammatoires rhumatismales ou intestinales. Les biopuces devraient contribuer à l'identification de cibles thérapeutiques pour la recherche pharmaceutique. Elles serviront aussi à déterminer la résistance aux antibiotiques de certaines souches microbiennes pour permettre de mieux lutter contre celles-ci.
La pharmacogénomique :
La
pharmacogénomique consiste à identifier les gènes
impliqués dans l'efficacité (ou l'inefficacité) d'un
produit, ou ses effets indésirables.
Elle conduit à une meilleure compréhension des mécanismes
d'action des médicaments. En montrant qu'une molécule a sur une
cible une action variable, la biopuce ouvre le champ des potentiels
thérapeutiques. Elle permet aussi d'identifier les effets secondaires
d'un produit et, lors des essais cliniques, de faire des mesures de
toxicité.
Le diagnostic des maladies infectieuses et génétiques
Ce
thème est développé dans le chapitre 1.2.4.
D'un point de vue économique, il est important de noter que le
marché mondial de diagnostic médical par biopuces sera en pleine
expansion d'ici quelques années.
En dehors du secteur médical, trois secteurs industriels non négligeables offrent des débouchés aux biopuces :
- L'agro-alimentaire : le suivi des bactéries
productrices de ferments lactiques, détection des séquences
provenant d'organismes génétiquement modifiés dans les
semences.
- L'environnement : l'analyse bactérienne de l'eau de consommation,
la détection des agents infectieux dans l'alimentation, l'air ou l'eau
(
Salmonella
,
Listeria
,
Legionnella
).
- La guerre bactériologique ou chimique : en déterminant par
avance les modifications du fonctionnement génétique des cellules
immunitaires occasionnées par des agents toxiques, on peut identifier
rapidement les produits chimiques (mercure, dioxine...) ou
bactériologiques (bacille du charbon ou de la diphtérie...)
disséminés par un éventuel agresseur.
Les multiples possibilités d'utilisation des biopuces expliquent que la
concurrence
règne entre les entreprises de biotechnologie qui
souhaitent dominer un
marché très rentable à moyen
terme
.
1.1.3.3. La production des biopuces
|
LES ACTEURS 18( * ) |
||||
|
|
SYNTENI |
AFFYMETRIX |
PROTOGENE |
|
|
Méthode de fabrication |
Dépôt de sondes PCR par robot à pointes |
Synthèse in situ par photolithographie |
Synthèse in situ par microfluidique |
|
|
Nature des sondes |
Sonde PCR : spécifique d'un gène |
Sonde " oligonucléotide " |
Sonde " oligonucléotide " |
|
|
Densité |
10 000 sondes/cm 2 |
100 000 sondes/cm 2 |
100-400 sondes/cm 2 |
|
|
Coût |
$ 4000/puce |
Programme de R & D : 2,2 M$ (1 seul design $450/puce) |
Prog. de R & D : 1,5 M$ (500 designs $300/puce) |
|
|
Applications |
Recherche Pharmacie |
Recherche |
Reséquençage Diagnostic |
|
|
|
40 000 gènes analysés par puce |
1700-7000 gènes analysés par puce |
1 gène analysé par puce (BRCA1, P53, HIV) |
|
Comme
dans bien d'autres domaines biotechnologiques, les États-Unis sont en
pointe pour les puces à ADN.
La société Affymetrix reste incontournable actuellement pour
plusieurs raisons :
Elle a mis au point la première puce à ADN et
bénéficie d'une expérience technologique certaine ;
Elle a protégé ses différentes techniques,
-notamment de dépôt de sondes sur la puce et de
séquençage par hybridation- par d'innombrables brevets. Ses
concurrents devront donc inévitablement la combattre
19(
*
)
, s'allier avec elle (c'est la
stratégie de Bio-Mérieux) ou tenter de la contourner en utilisant
des techniques différentes (c'est la solution adoptée par
Protogene).
Le fait que Glaxo-Welcome détienne 34 % du capital
d'Affymetrix lui confère une réelle solidité
économique.
Elle commercialise aussi les scanners de lecture automatique des biopuces
(près de 700 000 F le lecteur) à ceux qui achètent
ses biopuces et constituent en quelque sorte une clientèle captive.
Les biopuces sont aussi produites par d'autres sociétés
américaines, de moindre importance mais parfois associées
à de grands groupes de pharmacie ou d'instruments scientifiques :
Synteni (rachetée par Incyte Pharmaceuticals à la fin de 1997)
Hyseq (associée à Perkin Elmer), Molecular Dynamics, Nanogen, ...
En Europe, quelques équipes développent le procédé
des puces à ADN :
- l'Institut Engelhardt de Moscou travaille avec les groupes américains
Motorola et Packard Instrument, ainsi qu'avec le Laboratoire national d'Argonne
(Illinois).
- Le centre allemand de cancérologie de Heildelberg collabore avec
l'Université danoise de Copenhague.
- En Angleterre vient de voir le jour la société Oxford Gene
Technology, qui développe une technique particulière de fixation
des fragments d'ADN sur les puces.
- En France, la société CIS-Bio-International (Saclay)
étudie une technologie spécifique en collaboration avec le LETI
(Laboratoire d'électronique, de technologie et d'instrumentation) et le
DRFMC (département de recherche fondamentale de la matière
condensée) du CEA (Commissariat à l'énergie atomique)
à Grenoble.
Aujourd'hui, le LETI développe un nouveau programme : AMIGO,
exposé dans la deuxième partie du présent rapport.







